
Драфт этой статьи был написан почти два месяца назад. Затем я заболел ковидом, а потом долго восстанавливал свои когнитивные способности, изгоняя из себя древесину.
Предполагаю, что многие из нас видели фокусы. Волшебство. Когда хочется воскликнуть:
— Да как ты это сделал! Это же невозможно!
Давайте посмотрим, какие возможности предоставляет Houdini (софт назван в честь Гарри Гудини, да) для работы с разным количеством объектов/данных/точек. Где есть место фокусам. И как они могут помочь при работе с большими и тяжёлыми сценами. И не забываем, что наша конечная наша цель — визуальное представление этих данных.
Чем больше биологических объектов мы показываем, тем сложнее сцена. Один иммуноглобулин IgG – это 13 тысяч атомов. А если мы показываем вирус SARS-CoV-2, облепленный антителами и двигающийся по поверхности клетки, то это порядка 50 миллионов атомов.
Расшифрованные структуры всех белков (и не только), которые учёные хотят сделать общественным достоянием, передаются в глобальную базу — PDB (Protein Data Bank). И любой школьник/студент/аспирант/профессор/велосипедист может свободно использовать какую угодно структуру, ссылаясь на источник. Изумительный случай человеческого единства.
Данные хранятся в формате файла PDB/CIF. Это облако точек/атомов с координатами. Информации достаточно, чтобы описать структуру объекта в пространстве. Хотя данных там больше — есть информация о химических элементах, аминокислотах и прочем. Для нас сейчас важны только атомы и их координаты.
1 VDB (воксели/кубики)
Воксели позволяют представлять модель/точки в виде кубиков. Они могут быть больше или меньше. Воксели не используются в чистовом производстве, т.к. криволинейную поверхность (например, сферу) сложно представить кубами. Чтобы сгладить воксели их конвертируются в полигоны. Обратный процесс тоже возможен. Воксели занимают много места. При линейном увеличении размера объём вокселей увеличивается в кубической зависимости. Тогда зачем нам воксели?
Если у нас есть множество взаимно пересекающихся объектов, например, сферы, которыми мы изображаем атомы, то после конвертации в воксели мы получим однородную поверхность с единой плотностью и единой внешней поверхностью. Все внутренние пересечения будут удалены. Именно воксели лежат в основе всех молекулярных поверхностей, которые можно видеть на иллюстрациях PyMol и множестве другого софта. Также воксели используются в медицине (метод МРТ), при создании спецэффектов в кино и играх. Подробнее можно почитать в Википедии.
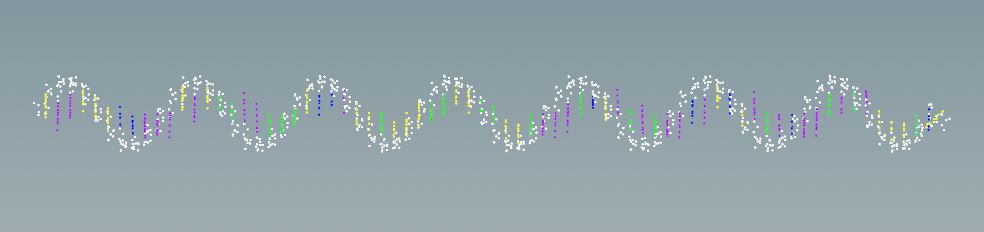
Мы видим небольшой фрагмент РНК в виде точек/атомов. Давайте преобразуем точки в VDB и чуть приблизимся. Что мы увидим?

Теперь конвертируем воксели в полигоны, подготавливаем материал (я выбрал красное стекло) и свет. Рендерим изображение.

Диаметры атомов усреднены. Модель чем-то похожа на оплывший воск или оплавленное цветное стекло, да. Прозрачность атомов, цвет, как и блики, разумеется, художественный вымысел.
Как я уже говорил, воксели очень требовательны к памяти и чем точнее нам нужно отобразить объект, тем меньше надо делать сам воксель, а значит, требование к памяти возрастает. Поэтому в чистом виде воксели используются с большой осторожностью.
Мы успешно преобразовали фрагмент РНК в воксели, а затем в полигоны. Полный геном РНК вируса SARS-CoV-2 (размер 30 тысяч нуклеотидов). Давайте посмотрим, как это выглядит.

Каждый нуклеотид на своём месте и каждый триплет кодирует нужную аминокислоту в структуре белка. Houdini пишет, что здесь 680 тысяч атомов. Давайте увеличим голубенький прямоугольник и посмотрим, что там.
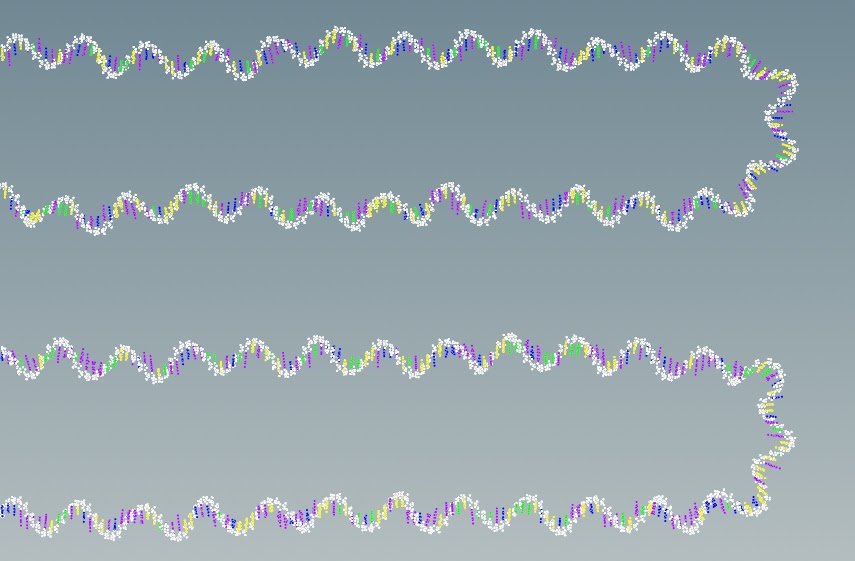
Цветом показаны азотистые основания и рибоза.
Мы видим, что это всё то же облако точек. Без сфер. Геометрически мы можем РНК уложить как угодно, что будет иметь мало общего с действительной укладкой РНК в вирусе. Гибкость укладки РНК средствами Houdini пригодится нам в дальнейшем работе с капсидом (внутренняя часть вируса). Представление данных в виде точек для нас фантастически удобно, т.к. занимает очень мало места в памяти, а значит, скорость работы с данными повышается. Такой способ хорош для представления данных на мониторе, но не в виде иллюстраций.

Воксели в полигоны мы можем конвертировать с разной степенью детализации, например, в зависимости от удалённости объекта от наблюдателя. Сделать изображение с полным РНК вируса в виде VDB, и уровнем детализации, как в средней части иллюстрации, потребует 50 гигабайт памяти. Что, разумеется, не выглядит хорошим решением, если у нас в сцене много объектов, а не только фрагмент РНК.
Хорошо. VDB отлично подходит для небольших объектов (молекулы иммуноглобулина или инсулина, к примеру). Как мы можем получить изображение с полным РНК вируса, если VDB эту задачу в лоб не решает или делает это с использованием очень больших ресурсов? В следующих частях я отвечу на этот вопрос.
2 Particles (частицы)
На помощь нам приходят частицы. Каждую точку из нашего облака точек мы можем представить в виде сферы. Но не на экране — это было бы очень ресурсозатратно. А на уровне рендера. На экране частицы так и останутся видны в виде точек. При этом размеры этих сфер, как и цвет, регулируемы. Более того, мы можем соотнести их с размерами углерода, кислорода и других химических элементов.
Давайте посмотрим, что в этом случае будет с РНК.

Мгновенный рендер, нетребовательный к памяти. 680 тысяч атомов.
Хорошо. Ведь это РНК вируса. А где он сам? Давайте на него посмотрим.
Ниже наша атомарная 3D модель вируса SARS-CoV-2. Более подробно об этом проекте можно почитать тут.
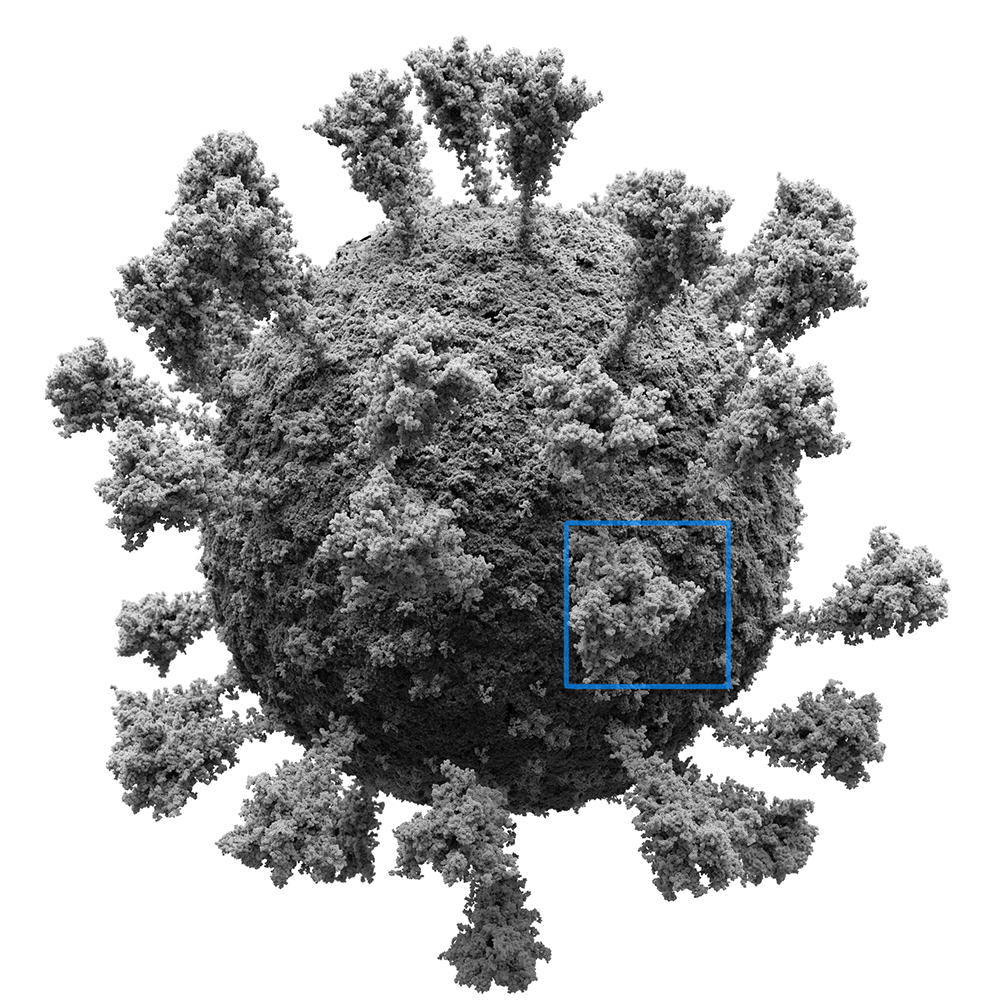
Вирус без капсида (внутренняя часть вируса). Здесь мы видим вирус целиком, но не видим насколько это детализированная модель. Давайте увеличим выделенный фрагмент.

Вот. Теперь мы видим атомы. Они также усреднены по размеру. В самом начале мы говорили, что в формат данных позволяет нам учесть химические элементы. В данном случае оставлен условный диаметр атома. И, если мы посмотрим на задний план, то увидим, что изображение нерезкое. Я добавил размытие/DOF, поэтому задний план в расфокусе.
Давайте посмотрим, что будет, если мы увеличим количество копий этой модели вируса.

Ох. При такой организации сцены 64 гигабайт памяти вмещают максимум 2 копии вируса. После этого Houdini говорит о нехватке памяти и прощается с нами. Почему так происходит? Давайте посмотрим, что мы можем с этим сделать.
3 Instance (копии с одинаковым серийным номером)
Визуально вирус состоит из одинаковых частей. Пожалуй, самые узнаваемые — S белки/шипы. В данной модели эти объекты различны, хотя и выглядят одинаково, как если бы каждый из шипов имел свой серийный номер. Если подумать, то в перспективе такой способ приводит к излишнему расходу памяти. Лайфхак: можно использовать объект с одним серийным номером, просто подставлять его в разные места. То же самое можно сказать и о копии целого вируса. Нам нужно сделать так, чтобы у нас была одна эталонная копия вируса, которую мы будем подставлять во все места, где будет вирус. Это позволит создать множество копий вируса. При этом память расходуется только на первую копию, а не на каждую.
По умолчанию в Houdini используется CPU рендер Mantra. Но есть возможность подключить другие внешние рендеры. Я использовал GPU рендер RedShift. Разница между AMD Ryzen 3950X (16 ядер) и NVIDIA GeForce 3080 RTX в моём случае была в семь раз в пользу видеокарты. Учитывая разнообразные тесты, а также работу с анимацией – очень существенная разница.
Проект делался в версии RedShift 3.0.35. В самой последней версии на сегодня 3.0.61 добавили поддержку RTX (аппаратная трассировка лучей). Выглядит это вот так.
С этой галочкой потенциально скорость рендера увеличится ещё в 3х раз. Это очень здорово! Но есть нюанс. Я установил последнюю версию RedShift. И эта версия оказалась несовместима со старыми камерами, светом и шейдерами. Поэтому для данного проекта я не могу использовать эту опцию и проверить её работоспособность. А вот в новых проектах – уже да.
Но, на всякий, случай ссылка, где тестировались GPU и CPU рендера в Houdini. И здесь видно, что GPU сильно предпочтительнее, если мы говорим о скорости рендера.
Итак. Нам понадобится нода instance. В которой мы указываем, какой объект мы копируем (вирус) и куда мы его копируем (облако точек). После выполнения каждая точка превратится в копию вируса. Память будет расходоваться только на эталонную копию вируса. Давайте посмотрим, что у нас получается.
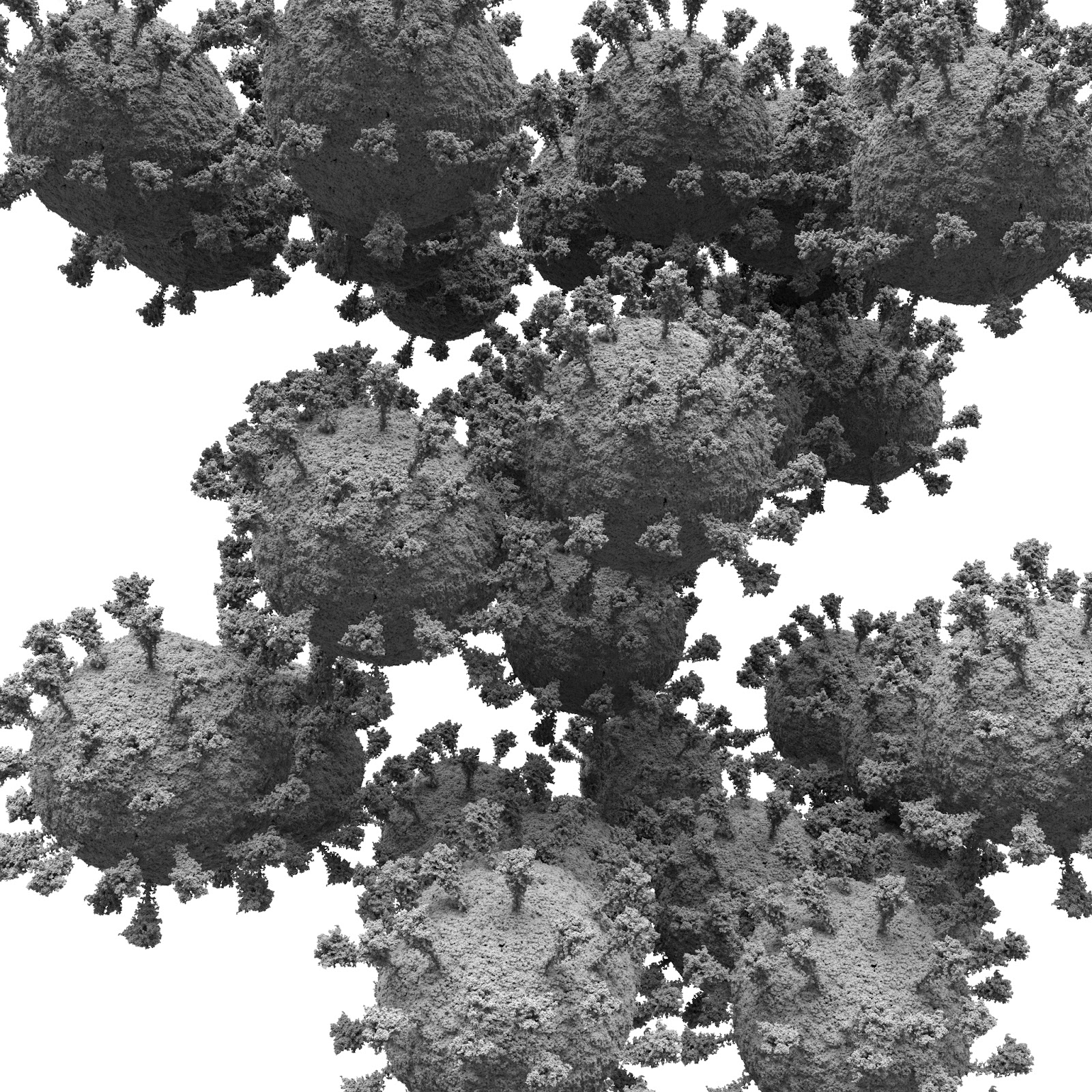
Миллиард! Теперь, потенциально, мы можем собирать сложные сцены, которые без этого инструмента на домашнем компьютере невозможны: проникновение множества копий вируса в клетку, ответ иммунной системы организма и прочее. В этом случае самыми ресурсоёмкими у нас будут первичные модели объектов, а вот количество копий уже будет несущественным. И это очень и очень здорово!
Хорошо. Но нам же не всегда нужны столь детализированная атомарная модель вируса. Если он очень далеко от наблюдателя, то мы не то, что атомы, мы и шипы-то его не увидим. Шарик и шарик. Можно что-то сделать с моделью, чтобы она не была столь тяжёлой?
4 LOD (level of detail – разные уровни детализации модели)
Да. На помощь нам приходит LOD.
Существующая модель вируса прекрасно подходит для крупных планов или деталей. Если вирус находится на заднем плане, то такая детализация избыточна – мы всё равно не увидим атомы, да и более крупные структуры тоже.
Если мы возьмём видео на youtube, vimeo, twitch, то это будет разрешение 1280х720, 1920х1080 и редко движение в сторону 2560 x 1440 и 3840 x 2160 пикселей/точек. Теперь, если мы перемножим ширину на высоту, то мы получим 1, 2, 4, 8 миллиона пикселей (округляем). Эти миллионы — максимальное количество пикселей для каждого кадра. Поэтому все наши миллиарды полигонов или точек превращаются в 1-8 миллионов пикселей. И чем больше разница между конечным форматом видео и тем количеством полигонов, которое мы хотим впихнуть, тем больше будет мерцание изображения от кадра к кадру. Поэтому идеально, когда задний план мы заменяем на текстуру.
Давайте посмотрим, что мы можем сделать, чтобы модель вируса занимала меньше места в памяти, не тормозила во вьюпорте, и не создавала проблем с мерцанием. А самое главное – визуально не отличалась от полноценной модели вируса.
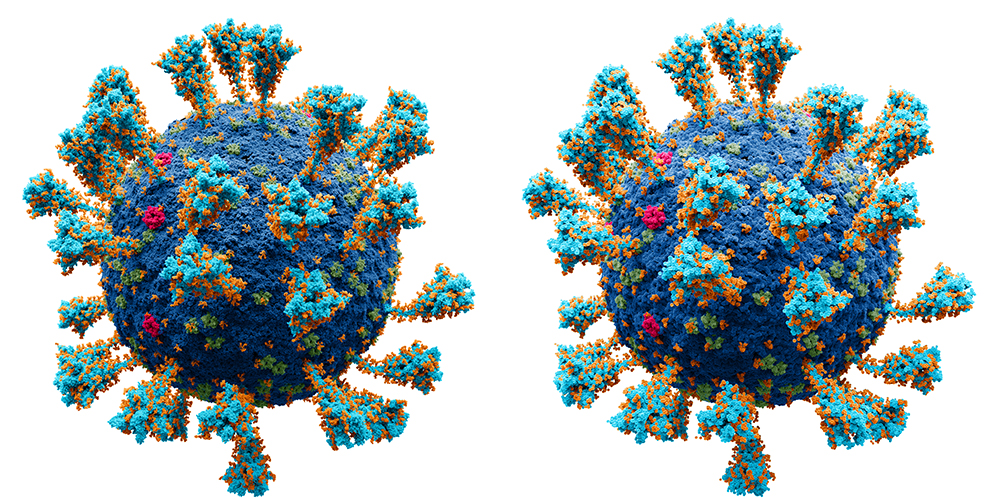
Это две разные модели одного и того же вируса. Визуально, в таком ракурсе обе эти иллюстрации идентичны. Для нас это означает, что облегчённая версия великолепно справляется со своей задачей. Давайте приблизимся поближе.
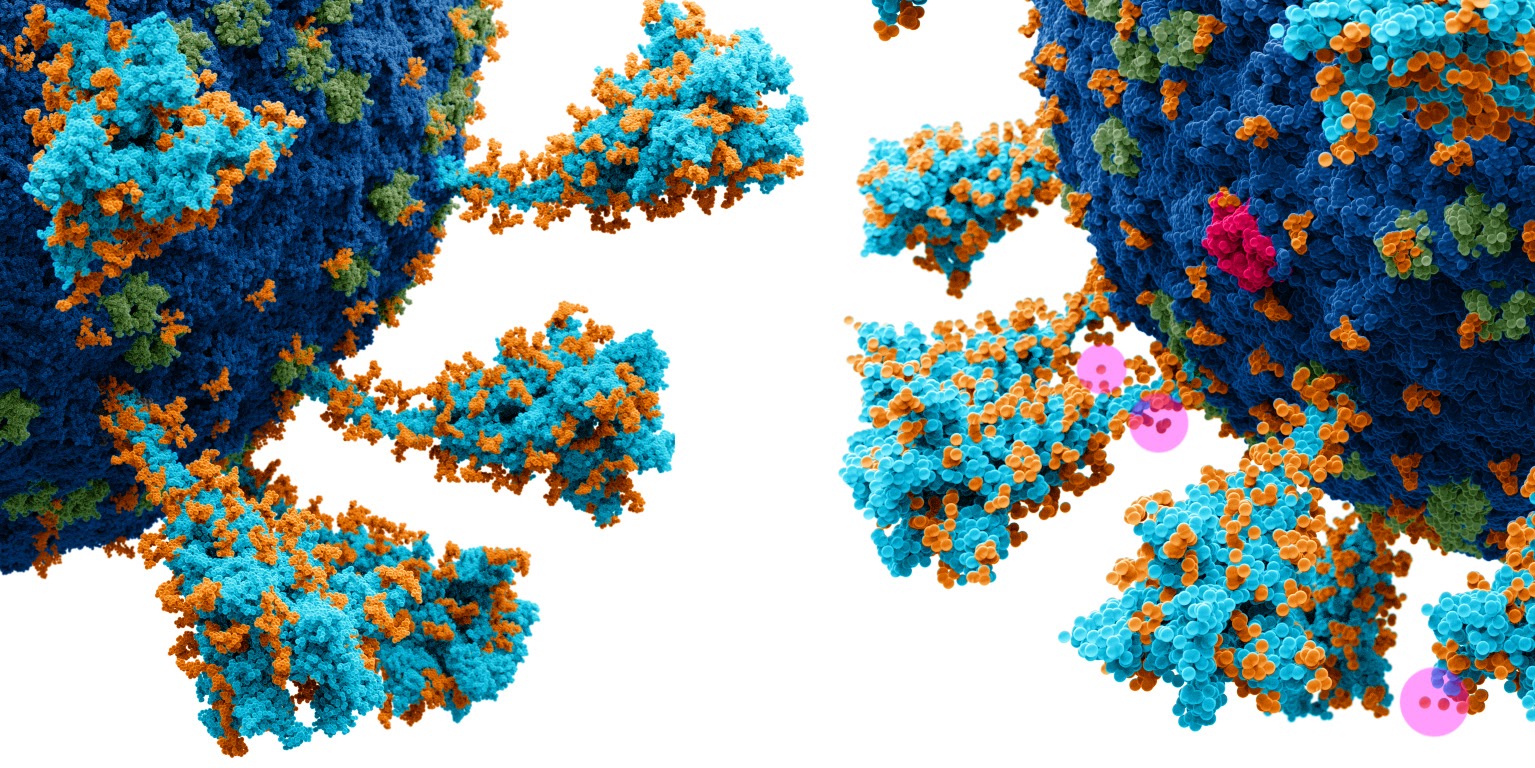
Разрешающей способности модели слева достаточно и для больших разрешений. А вот у модели справа видны оторвавшиеся атомы. Они висят в воздухе. Поэтому и визуальной, и с научной точки зрения, такая модель не годится. Не приближаясь так близко, мы делаем модель жизнеспособной. Помните про самолёт в виде точки?
И теперь мы можем использовать для переднего плана одну модель, а для задних планов – другую. Разумеется, можно создать и промежуточные варианты. Более того — нашу облегчённую модель сделать ещё легче, например, в виде шарика и палочек. Почему нет?
Хорошо. Мы увидели разные по уровню детализации модели. И увидели их возможности и ограничения. Но как они были получены? За счёт чего?
5 Тесселяция (разбиение полигонов)
Переразбиение полигонов модели с целью уменьшить их количество. Давайте посмотрим, как это работает.

Чтобы получить три варианта полигональной модели шип коронавируса, мы сначала преобразовали облако атомов из pdb файла в VDB, и уже потом в полигоны с разной степенью детализации. Мы уменьшили на порядок количество полигонов в модели. Без визуально ощутимой потери в качестве. Безусловно, для нас важно, насколько вблизи мы смотрим на модель. И самая лёгкая модель на ближних ракурсах будет смотреться хуже, чем на дальних.
Вот как выглядит модель, если мы приблизимся и посмотрим на фрагмент без материала.

Безусловно, такой инструмент позволяет нам и уменьшить размер модели в памяти и работать с большим количеством объектов во вьюпорте. Хорошо, это полигоны. А как обстоят дела с частицами/точками?
В этом случае всё не столь радужно. Давайте посмотрим, что получается.

В данном случае атомы буквально удалены. А это значит, что химические связи разорваны. И фактически такое положение атомов не будет удерживаться в виде геометрии S-шипа. Атомы разлетятся в поиске новых химических связей. Но мы можем проделать такую операцию, т.к. наша цель — иллюстрация. И если представить, что мы рисуем самолёт, то я нарисовал его в виде точки. Без попытки вместить туда иллюминаторы, шасси и пассажиров.
Средний вариант на иллюстрации выше вполне хорош. А вот крайний совсем не годится. Атомы отрываются друг от друга. Что делать?
Если мы постепенно будем увеличивать диаметр сфер, то тогда эту задачу получится решить. Выглядеть это будет вот так.

В этом случае мы игнорируем радиусы атомов, как и ван-дер-ваальсовые радиусы, т.к. объект будет на таком удалении, что ничего этого мы не увидим. Вспоминаем про самолёт.
Разумеется, в этом случае нам нужно следить, чтобы у остальных частей вируса диаметры (фактически условные размеры атомов) были такими же.
В случае с частицами мы удаляем точки/атомы. Поэтому мы теряем в научной точности представления модели. Что очень хорошо видно, если мы смотрим на модель вблизи. Именно удалённость модели от наблюдателя/камеры позволяет нам пренебречь этим фактом — мы всё равно не увидим этих удалённых атомов.
Эти одинокие атомы — результат несовершенств методов определения структуры белковых молекул (рентгеноструктурный анализ, криоэлектронная микроскопия, ядерный магнитный резонанс и прочее)
6 Отсечение геометрии по камере
Сцена обычно состоит из бОльшего числа объектов, чем видит камера/наблюдатель. У нас есть объекты, которые мы никогда не увидим. Но они, разумеется, занимают ресурсы в памяти и сильно мешают при работе со сценой.

На финальном ракурсе анимации мы видим вирус, вдалеке ещё одну копию вируса, поверхность клетки и цветом выделены рецептор АСЕ2 и S-шип. В таком ракурсе мы не увидим капсид (внутреннюю часть вируса). Поэтому их можно не показывать. Если продолжить эту мысль, то мы не увидим и всё, что скрывает обратная сторона вируса. А ещё мы не увидим такой же вирус, который находится за наблюдателем/камерой. Часть мембраны клетки слева и справа мы тоже не увидим. Давайте посмотрим, что мы можем сделать с этими лишними объектами.

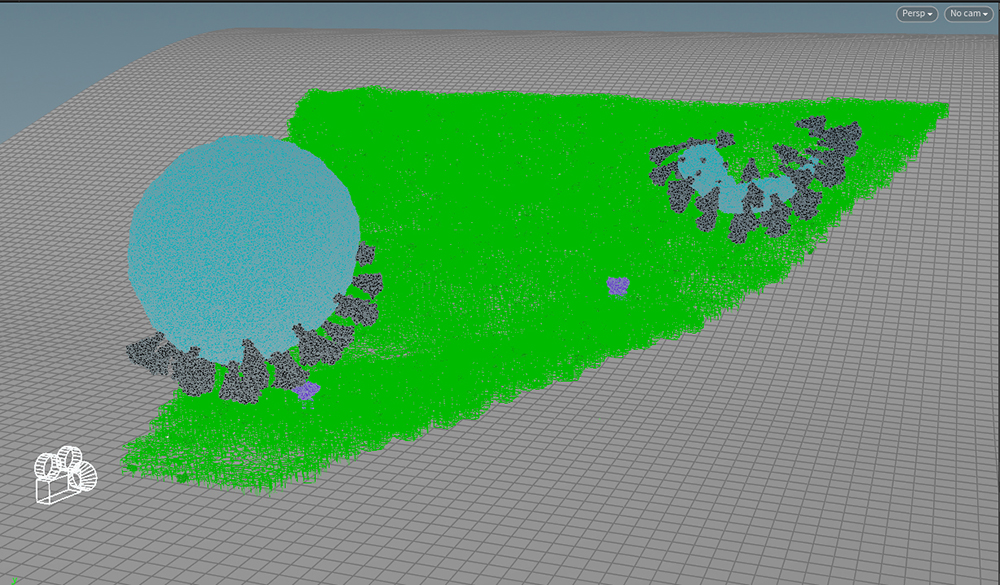
А вот так выглядит сцена на самом деле. Это похоже на онлайн-конференции, когда солидность видимой части участника нивелируется пижамными штанами, которые в камеру не попадают. Мы видим сильно облысевшие копии вирусов. Один из них даже утратил часть своей мембраны. Зелёный цвет – фрагмент мембраны клетки, который попадает в камеру. Именно он “заселяется” липидами и рецепторами. Весьма существенная разница, правда?
7 Итого
Мы рассмотрели ряд способов работы с атомарными данными для молекулярной биологии. Начали с 2 тысяч атомов и закончили на сцене в 1 миллиард атомов. И увидели, что если задачу решать в лоб/честно, то ресурсы заканчиваются очень быстро. Houdini учит нас иллюзии/фокусам, как сохранить ощущение волшебства, иллюзорность подлинности. И именно это позволяет нам двигаться дальше и увеличивать количество атомов на порядки. Мы рассмотрели не все возможности, которые потенциально есть в Houdini для решения схожих задач, а только те, что использовались в наших проектах.
Все изображения получены мной или в соавторстве с Валерией Архиповой. Изображения создавались в Houdini, с использованием GPU рендера RedShift и видеокарты NVIDIA GeForce 3080 RTX.
В работе использованы белки из PDB: 6vsb, 2mls, 5x29, 6yyt.
Алексей Солодовников (Houdini artist)
Валерия Архипова (Molecular biologist)